兩組獨立樣本變異數相同 t 檢定 (Two-Sample t test with equal variances,parametric)
套路 9: 兩組獨立樣本變異數相同 t 檢定
(Two-Sample t test with
equal variances,parametric)
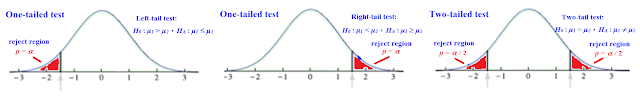
什麼是兩組獨立樣本假設檢定? 說白了就是兩組分別獨立取樣的資料做比較的假設檢定。統計假設檢定檢定什麼?看H0。例如兩組獨立樣本假設檢定H0 : μ1
= μ2,HA : μ1
¹ μ2是檢定兩組資料的平均值是否相同。又例如兩組獨立樣本假設檢定H0 : μ1 < μ2,HA : μ1
³ μ2是檢定第一組資料的平均值是否小於第二組資料的平均值。假設相等時為雙尾 (two-tailed test) 檢定。假設不相等時為單尾 (one-tailed test) 檢定。如下圖所示:
1. 使用時機: 用於比較兩組變異數相同獨立樣本的平均值(mean)。大樣本用Z檢定小樣本用t檢定。
2. 分析類型: 母數分析(parametric analysis)。直接使用資料數值算統計叫parametric方法,把資料排序之後用排序的名次算統計叫non-parametric方法。
3. 前提假設: 資料為常態分布(normal distribution) 或接近常態分布,變異數相同(equal variances)。
4. 資料範例: 咪路調查高一和大一學生體重(kg),資料如下:
|
高一
|
41
|
35
|
33
|
36
|
40
|
46
|
31
|
37
|
34
|
30
|
38
|
|
大一
|
52
|
57
|
62
|
55
|
64
|
57
|
56
|
55
|
60
|
59
|
|
請問平均體重是否相同? H0: m1 = m2,HA: m1 ≠ m2。
5. 畫圖看資料分布:
wt = [41, 35, 33, 36, 40, 46, 31, 37,
34, 30, 38, 52, 57, 62, 55, 64, 57, 56, 55, 60, 59]
cl =
["H","H","H","H","H","H","H","H","H","H","H","U","U","U","U","U","U","U","U","U","U"]
dat = {'Weight':wt,'Class':cl}
import pandas as pd
df = pd.DataFrame(dat)
import seaborn as sns
sns.set(style="whitegrid")
ax = sns.boxplot(x =
"Class", y = "Weight", data = df, width=0.2,
palette="Set3")
ax = sns.swarmplot(x =
"Class", y = "Weight", data = df, color = "red")
結果:
6. 檢查資料是否為常態分布 (H0:資料為常態分佈):
dat1 = [41, 35, 33, 36, 40, 46, 31,
37, 34, 30, 38]
dat2 = [52, 57, 62, 55, 64, 57, 56,
55, 60, 59]
import scipy.stats
scipy.stats.shapiro(dat1)
結果: (0.9705673456192017,
0.8921969532966614)
p = 0.8922 > 0.05,接受H0:資料為常態分佈。
scipy.stats.shapiro(dat2)
結果: (0.9728113412857056,
0.9156168699264526)
p = 0.9156 > 0.05,接受H0:資料為常態分佈。
7. 檢查資料是否為相同變異數 (H0: s12 = s22):
dat1 = [41, 35, 33, 36, 40, 46, 31,
37, 34, 30, 38]
dat2 = [52, 57, 62, 55, 64, 57, 56,
55, 60, 59]
import scipy.stats
scipy.stats.levene(dat1, dat2, center
= 'mean')
結果: LeveneResult(statistic=0.492665294888687,
pvalue=0.4912503045382468)
p = 0.491 > 0.05,接受H0: s12 = s22。
# 相同變異數表示樣本來自相同母體(population),不同變異數表示樣本取樣自不同母體。
8. 使用Python計算兩組獨立樣本變異數相同t檢定:
方法一: Python函數
dat1 = [41, 35, 33, 36, 40, 46, 31, 37,
34, 30, 38]
dat2 = [52, 57, 62, 55, 64, 57, 56,
55, 60, 59]
import scipy.stats
scipy.stats.ttest_ind(dat1,
dat2, equal_var = True)
結果: Ttest_indResult(statistic=-11.582948095369163,
pvalue=4.697622207379728e-10)
p = 4.697e-10 < 0.05,不接受假設H0: m1 = m2,平均體重不相同。
方法二: Python 函數
dat1 = [41, 35, 33, 36, 40, 46, 31,
37, 34, 30, 38]
dat2 = [52, 57, 62, 55, 64, 57, 56,
55, 60, 59]
import statsmodels.stats.weightstats
d1 =
statsmodels.stats.weightstats.DescrStatsW(dat1)
d2 = statsmodels.stats.weightstats.DescrStatsW(dat2)
statsmodels.stats.weightstats.CompareMeans(d1,
d2).ttest_ind(alternative = 'two-sided', usevar = 'pooled', value = 0)
# 相同變異數: usevar = 'pooled',不同變異數: usevar = 'unequal'。 value = 上述t公式中m1 - m2的值。
結果: (-11.582948095369163,
4.697622207379728e-10, 19.0)
p = 4.697e-10 < 0.05,不接受假設H0: m1 = m2,平均體重不相同。





留言
張貼留言